Identificación de un fragmento de la región del espaciador interno transcrito del ADNr en Cenchrus purpureus (Poaceae)
Contenido principal del artículo
Resumen
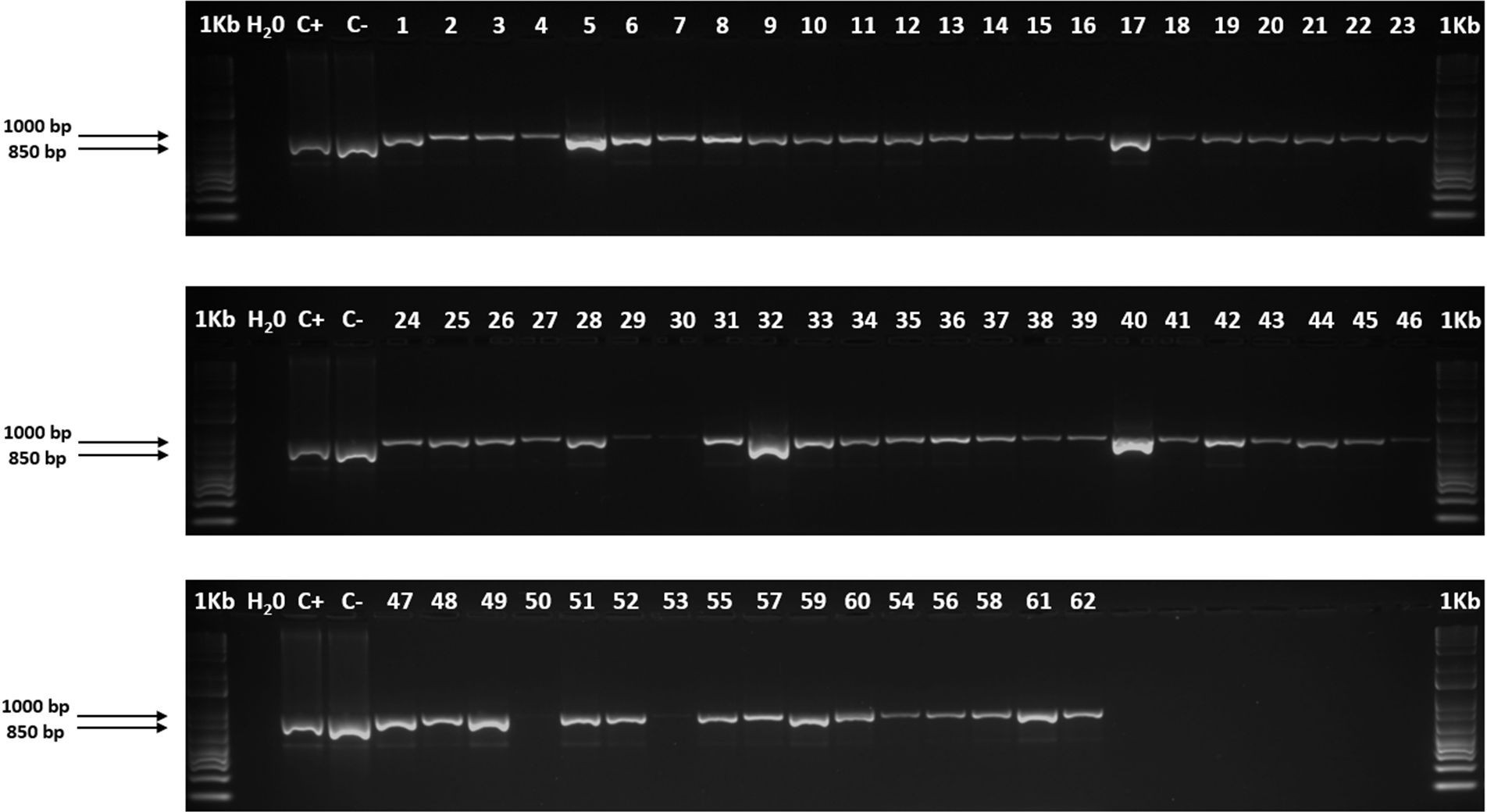
La región del espaciador interno transcrito (ITS), es fácil de amplificar por sus numerosas copias presentes en el genoma vegetal, pero no se reportan investigaciones de esta región en la especie forrajera Cenchrus purpureus. El presente estudio tuvo como objetivo identificar mediante Reacción en Cadena de la Polimerasa (PCR) un fragmento de la región ITS del ADN ribosomal del núcleo en C. purpureus mediante los cebadores ITS1/ ITS4 y compararlo con dos muestras de Urochloa spp. En el estudio molecular se emplearon muestras de ADN de 62 accesiones de C. purpureus del banco de germoplasma de pastos y forrajes, pertenecientes al Instituto de Ciencia Animal y dos muestras de accesiones de Urochloa spp. del Programa de Recursos Genéticos de la Alianza de Bioversity International y el Centro Internacional de Agricultura Tropical. El ADN de las 62 accesiones de C. purpureus y muestras de Urochloa spp., amplificaron con los cebadores ITS1/ITS4. Los productos de amplificación revelaron una banda polimórfica nítida en los geles para cada accesión y tamaño aproximado de 1000 pb en C. purpureus y 850 pb en Urochloa spp. Los cebadores determinaron diferencias en las amplificaciones entre las muestras de C. purpureus y Urochloa spp. aunque no demostraron variabilidad entre accesiones de la misma especie. Estos marcadores moleculares se pueden utilizar para comprobar la amplificación, mediante PCR, de muestras de ADN de C. purpureus y en la diferenciación de especies en géneros de Poaceae.
Detalles del artículo

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Aquellos autores/as que tengan publicaciones con esta revista, aceptan los términos siguientes:
- Los autores/as conservarán sus derechos de autor y garantizarán a la revista el derecho de primera publicación de su obra, el cuál estará simultáneamente sujeto a la Licencia Creative Commons Attribution-NonCommercial 4.0 International (CC BY-NC 4.0) que permite a terceros compartir la obra siempre que se indique su autor y su primera publicación esta revista. Bajo esta licencia el autor será libre de:
- Compartir — copiar y redistribuir el material en cualquier medio o formato
- Adaptar — remezclar, transformar y crear a partir del material
- El licenciador no puede revocar estas libertades mientras cumpla con los términos de la licencia
Bajo las siguientes condiciones:
- Reconocimiento — Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.
- NoComercial — No puede utilizar el material para una finalidad comercial.
- No hay restricciones adicionales — No puede aplicar términos legales o medidas tecnológicas que legalmente restrinjan realizar aquello que la licencia permite.
- Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
- Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos telemáticos institucionales o en su página web) antes y durante el proceso de envío, lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).
Citas
Ahmadi, H., Solouki, M., Fazeli-Nasab, B., Heidari, F. & Sayyed, R.Z. 2022. Internal transcribed spacer (ITS) regions: A powerful tool for analysis of the diversity of wheat genotypes. Indian Journal of Experimental Biology, 60(2): 137-143, ISSN: 0975-1009. http://op.nis- cair.res.in/index.php/IJEB/article/view/34886.
Al Rahbawi, S.M., Al-Edhari, A.H. & Sardar, A.S. 2021. Phylogenetic Study of the Genus Phalaris L. (Poaceae) based on Nuclear Internal Transcript Region (ITS) in Iraq. Annals of the Romanian Society for Cell Biology, 25(3): 8278-8281, ISSN: 2067-8282. http://annalsofrscb.ro/in- dex.php/journal/article/view/2368.
Alaklabi, A. 2021. Ficus Species Genetic Diversity Based on Internal Transcribed Spacer (ITS) Region Analysis. Egyptian Academic Journal of Biological Sciences, H. Botany, 12(1): 21-27, ISSN: 2090-3820. https://doi.org/10.21608/eajbsh.2021.148134.
Almutairi, Z.M. 2021. Molecular identification and phylogenetics of local pearl millet cultivars using internal-transcribed spacers of nuclear ribosomal DNA. Plant Genetic Resources: Characterization and Utilization, 19(4): 339-346, ISSN: 1479-2621. https://doi.org/10.1017/S1479262121000393.
Álvarez Báez, Y. 2021. Comportamiento productivo de nuevas variedades de Cenchrus purpureus en el Valle del Cauto, Cuba. Tesis de Doctorado. Universidad de Granma. Cuba. p. 99.
Álvarez, Y., Herrera, R.S., Ramírez, J.L., Verdecia, D.M., Benítez, D. & López, S. 2024. Performance of Cenchrus purpureus varieties tolerant to salinity under the edaphoclimatic conditions of Granma province, Cuba. Cuban Journal of Agricultural Science, 58: e01, ISSN: 2079-3480. https://cu-id.com/1996/v58e01.
Baldwin, B.G., Sanderson, M.J., Porter, J.M., Wojciechowski, M.F., Campbell, C.S. & Donoghue, M.J. 1995. The ITS region of nuclear ribosomal DNA: a valuable source of evidence on angiosperm phylogeny. Annals of the Missouri Botanical Garden, 82(2): 247-277, ISSN: 2162-4372.
https://www.jstor.org/stable/2399880.
Bult, C. & Zimmer, E. 1993. Nuclear ribosomal RNA seguences for inferring tribal relationships within Onagraceae. Systematic Botany, 18(1): 48-63, ISSN:
-2324. https://www.jstor.org/stable/2419787.
Chen, Z.T., Huang, Q.L., Pan, W.B. & Huang, Y.B. 2010. Sequence analysis of the rDNA ITS region of Pennisetum species (Poaceae). Acta Prataculturae Sinica, 19(4): 135, ISSN: 1004-5759. http://cyxb.magtech.com.cn/EN/Y2010/V19/I4/135.
Cruz, R., Sosa, A., Herrera, R.S. & Martínez, R.O. 1993. Identificación electroforética de Pennisetum purpureum cv. King grass. Revista Cubana de Ciencia Agrícola, 27(2): 219-223, ISSN: 2079-3480. https://cjascience.com/in-dex.php/CJAS.
de Vere. N., Rich, T.C.G., Ford, C.R., Trinder, S.A., Long, C., Moore, C.W., Satterthwaite, D., Davies, H., Allainguillaume, J., Ronca, S., Tatarinova, T. Garbett, H., Walker, K. & Wilkinson, M.J. 2012. DNA Barcoding the Native Flowering Plants and Conifers of Wales. PLoS ONE, 7(6): e37945, ISSN: 1932-6203. https://doi.org/10.1371/journal.pone.0037945.
Fortes, D., Herrera, R.S. & Herrera, M. 2023. Morphoagronomic performance of Cenchrus purpureus new clones. Cuban Journal of Agricultural Science, 57: 8, ISSN: 2079-3480. https://cjascience.com/in-dex.php/CJAS/article/view/1105.
Ghosh, J. S., Bhattacharya, S. & Pal, A. 2017. Molecular phylogeny of 21 tropical bamboo species reconstructed by integrating non-coding internal transcribed spacer (ITS1 and 2) sequences and their consensus secondary structure. Genetica, 145(3): 319-333, ISSN: 1573-6857. https://doi.org/10.1007/s10709-017-9967-9.
González, A.C. 2002. Detección del polimorfismo genético mediante marcadores bioquímicos en plantas. En: Marcadores moleculares: nuevos horizontes en la genética y la selección de las plantas. Cornide, M. T. (Coord.). Ed. Félix Varela, La Habana, Cuba. pp. 36-63. ISBN: 959-258-351-X.
González, A.T. & Morton, C.M. 2005. Molecular and morphological phylogenetic analysis of Brachiaria and Urochloa (Poaceae). Molecular Phylogenetics and Evolution, 37(1): 36-44, ISSN: 1095-9513. https://doi:10.1016/j.ympev.2005.06.003.
González, C. & Martínez, R.O. 2019. Genetic characterization of clones and varieties of Cenchrus purpureus with microsatellite markers. Cuban Journal of Agricultural Science, 53(3): 307-318, ISSN: 2079-3480. https://cjas-cience.com/index.php/CJAS/article/view/914.
Herrera, R., García, M. & Cruz, A.M. 2019. Study of morphoagronomic indicators of Cenchrus purpureus clones for biomass production. Cuban Journal of Agricultural Science, 53(2): 189-196, ISSN: 2079-3480. https://cjascience.com/index.php/CJAS/article/view/886.
Herrera, R.S. 2022. Evaluation of Cenchrus purpureus varieties tolerant to drought in the western region of Cuba. Cuban Journal of Agricultural Science, 56(2): 135-143, ISSN: 2079-3480. https://cjascience.com/in-dex.php/CJAS/article/view/1049.
Hsiao, C., Chatterton, N. J., Asay, K. H. & Jensen, K. B. 1995. Molecular phylogeny of the Pooideae (Poaceae) based on nuclear rDNA (ITS) sequences. Theoretical and Applied Genetics, 90: 389-398, ISSN: 1432-2242. https://doi.org/10.1007/BF00221981.
Hsiao, C., Jacobs, S.W.L., Chatterton, N.J. & Asay, K.H. 1998. A molecular phylogeny of the grass family (Poaceae) based on the sequences of nuclear ribosomal DNA (ITS). Australian Systematic Botany, 11(6): 667-688, ISSN:
-5701. https://doi.org/10.1071/SB97012.
Jones, L., Twyford, A.D., Ford, C.R., Rich, T.C., Davies,
H., Forrest, L.L., Hart, M.L., McHaffie, H., Brown, M.R., Hollingsworth, P.M. & De Vere, N. 2021. Barcode UK: A complete DNA barcoding resource for the flowering plants and conifers of the United Kingdom. Molecular Ecology Resources, 21(6): 2050-2062. ISSN: 1755-0998.
https://doi.org/10.1111/1755-0998.13388.
Liao, H.C., Ming-Hui, C. & Chih-Hui, C. 2020. Barcode of nuclear ribosomal internal transcribed spacer regions (ITS) as a useful tool to recognize a newly naturalized and potentially invasive weed, Chloris pilosa Schumach. (Poaceae), in Taiwan. Taiwania, 65(2): 129, ISSN: 0372-333X. https://doi.org/10.6165/tai.2020.65.129.
Liston, A., Robinson, W.A., Oliphant, J.M. & Álvarez-Buylla,
E.R. 1996. Length variation in the nuclear ribosomal DNA internal transcribed spacers of non-flowering seed plants. Systematic Botany, 21(2): 109-120, ISSN: 1548-2324. https://doi.org/10.2307/2419742.
Nadeem, M.A., Nawaz, M.A., Shahid, M.Q., Doğan, Y., Comertpay, G., Yıldız, M., Hatipoğlu, R., Ahmad, F., Alsaleh, A., Labhane, N., Özkan, H., Chung, G. & Baloch, F.S. 2018. DNA molecular markers in plant breeding: current status and recent advancements in genomic selection and genome editing. Biotechnology & Biotechnological Equipment, 32(2): 261-285, ISSN: 1310-2818. https://doi.org/10.1080/13102818.2017.1400401.
Nguyen, B.T., Le, L.B., Pham, L.P., Nguyen, H.T., Tran,
T.D. & Van Thai, N. 2021. The effects of biochar on the biomass yield of elephant grass (Pennisetum purpureum Schumach) and properties of acidic soils. Industrial Crops and Products, 161: 113224, ISSN: 1872-633X. https://doi.org/10.1016/j.indcrop.2020.113224.
Özbek, Ö. 2024. Molecular Markers Used to Reveal Genetic Diversity and Phylogenetic Relationships in Crop Plants. OBM Genetics, 8(4): 1-25, ISSN: 2577-5790. https://doi.org/10.21926/obm.genet.2404274.
POWO. 2025. Plants of the World Online. Kew Royal Botanic Gardens. Available at: https://powo.sci-ence.kew.org/taxon/urn:lsid:ipni.org:names:77106033-1. [Consulted: January 03, 2025].
Risterucci, A., Grivet, L., N’Goran, Pieretti, J., Flament, M. & Lanaud, C. 2000. A high-density linkage map of Theobroma cacao L. Theoretical and Applied Genetics, 101: 948-955, ISSN: 0040-5752. https://doi.org/10.1007/s001220051566.
Sun, Y., Skinner, D.Z., Liang, G.H. & Hulbert, S.H. 1994. Phylogenetic analysis of Sorghum and related taxa using internal transcribed spacers of nuclear ribosomal DNA. Theoretical and Applied Genetics, 89: 26-32, ISSN: 1432-2242. https://doi.org/10.1007/BF00226978.
Tan, F., He, L., Zhu, Q., Wang, Y., Chen, C. & He, M. 2022. Pennisetum hydridum: a potential energy crop with multiple functions and the current status in China. BioEnergy Research, 15: 850-862, ISSN: 1939-1242. https://doi.org/10.1007/s12155-021-10263-7.
Wang, J., Yan, Z., Zhong, P., Shen, Z., Yang, G. & Ma, L. 2022. Screening of universal DNA barcodes for identifying grass species of Gramineae. Frontiers in Plant Science, 13: 998863, ISSN: 1664-462X. https://doi.org/10.3389/fpls.2022.998863.
Wessapak, P., Ngernsaengsaruay, C. & Duangjai, S. 2023. A taxonomic revision of Cenchrus L. (Poaceae) in Thailand, with lectotypification of Pennisetum macrostachyum Benth. PhytoKeys, 234: 1-33, ISSN: 1314-2003. https://doi.org/10.3897/phytokeys.234.106486.
White, T. J., Bruns, T., Lee, S.J.W.T. & Taylor, J. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: PCR protocols: a guide tomethods and applications. M. Innis, D. Gelfand, J. Sninsky & T. White (eds.). Academic Press, INC. San Diego California. pp. 315-322. ISBN: 0-12-372181-4.